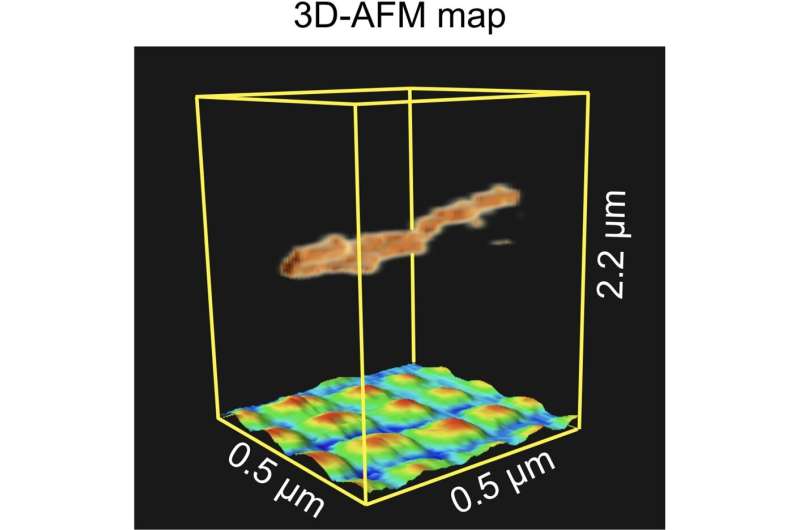
Driedimensionale atomic force microscopy (3D-AFM) kaart die de mogelijkheid onderstreept om zwevende flexibele monsters af te beelden boven een regelmatig patroon van nano-puntjes. Credit: Kleine methoden (2024). DOI: 10.1002/smtd.202400287
Onderzoekers van het Nano Life Science Institute (WPI-NanoLSI), Kanazawa University, rapporteren de 3D-imaging van een zwevende nanostructuur. De gebruikte techniek is een uitbreiding van atomaire krachtmicroscopie en is een veelbelovende benadering voor het visualiseren van verschillende 3D-biologische systemen.
Atomic force microscopy (AFM) werd oorspronkelijk uitgevonden om oppervlakken te visualiseren met nanoschaalresolutie. Het basisprincipe is om een ultradunne punt over het oppervlak van een monster te bewegen. Tijdens deze xy-scanbeweging volgt de positie van de punt in de richting loodrecht op het xy-vlak het hoogteprofiel van het monster, wat resulteert in een hoogtekaart van het oppervlak.
De laatste jaren zijn er manieren verkend om de methode uit te breiden naar 3D-beeldvorming, waarbij onderzoekers van het Nano Life Science Institute (WPI-NanoLSI) van de Kanazawa University baanbrekende experimenten met levende cellen rapporteerden. Om 3D-AFM echter te laten evolueren tot een breed toepasbare techniek voor het visualiseren van flexibele moleculaire structuren, is een grondig begrip van de beeldvormingsmechanismen die hierbij een rol spelen, noodzakelijk.
Nu hebben Takeshi Fukuma van de Kanazawa University en collega’s een gedetailleerde studie uitgevoerd van een speciaal ontworpen flexibel monster, wat essentiële inzichten biedt in de theoretische basis en de interpretatie van 3D-AFM-experimenten. De studie is gepubliceerd in het dagboek Kleine methoden.
Met behulp van microfabricatietools creëerden de wetenschappers een monster bestaande uit een koolstofnanobuisvezel die rust op platinapilaren die op hun beurt op een siliciumsubstraat waren geplaatst. Een koolstofnanobuis is een structuur die men kan zien als een opgerolde, één atoom dikke koolstofplaat. Het vrijstaande deel van de nanobuis was ongeveer 2 micrometer lang. De hele structuur werd ondergedompeld in water, aangezien veel 3D biomoleculaire systemen van belang voorkomen in vloeibare omgevingen.
Fukuma en collega’s voerden vervolgens 3D-AFM-experimenten uit in twee verschillende modi. In de statische modus wordt de nanotip verticaal naar het monster verlaagd. Wanneer de tip contact maakt met de zwevende nanotube-vezel, wordt deze opzij geduwd en buigt terwijl de sonde verder daalt. In de dynamische modus wordt de tip, die aan een cantilever is bevestigd, op een resonantiefrequentie laten oscilleren terwijl hij wordt verlaagd.
Door te analyseren hoe de kracht die de punt ervaart verandert als functie van de diepte van de punt, concludeerden de onderzoekers dat de wrijving tussen de punt en de vezel veel groter is in de statische modus vergeleken met de dynamische modus. De laatste is daarom de modus van keuze, omdat minder wrijving betekent dat potentiële schade aan het monster minder waarschijnlijk is.
De wetenschappers voerden computersimulaties uit om te modelleren wat er gebeurt als de punt de koolstofnanotubevezel bereikt. De simulaties bevestigden dat de zwevende nanotube lateraal verplaatst en dat een continu trillende punt (zoals in de dynamische modus) resulteert in zwakkere krachten die door het monster worden ervaren, waardoor sterke hechting van de punt aan de vezel wordt belemmerd.
Fukuma en collega’s voerden vervolgens experimenten uit met een koolstofnanobuisvezel die boven een regelmatig patroon van nano-sized platina dots was gehangen die op een siliciumsubstraat waren afgezet. De metingen werden in dynamische modus uitgevoerd. De gereconstrueerde 3D-kaart van het gescande volume toonde duidelijk de vezel en de dots eronder, wat de mogelijkheid van 3D-AFM om verticaal overlappende nanostructuren af te beelden, onderstreepte.
Deze bevindingen tonen aan dat AFM over het algemeen kan worden toegepast om flexibele 3D-structuren te visualiseren. “De vorderingen die in deze studie zijn gemaakt, kunnen mogelijk leiden tot meer gedetailleerde en nauwkeurige AFM-analyse van verschillende 3D-biologische systemen zoals cellen, organellen, chromosomen en vesikels”, stellen de wetenschappers.
Meer informatie:
Mohammad Shahidul Alam et al, Onthulling van het mechanisme dat ten grondslag ligt aan 3D-AFM-beeldvorming van zwevende structuren door experimenten en simulaties, Kleine methoden (2024). DOI: 10.1002/smtd.202400287
Informatie over het tijdschrift:
Kleine methoden
Aangeboden door Kanazawa University