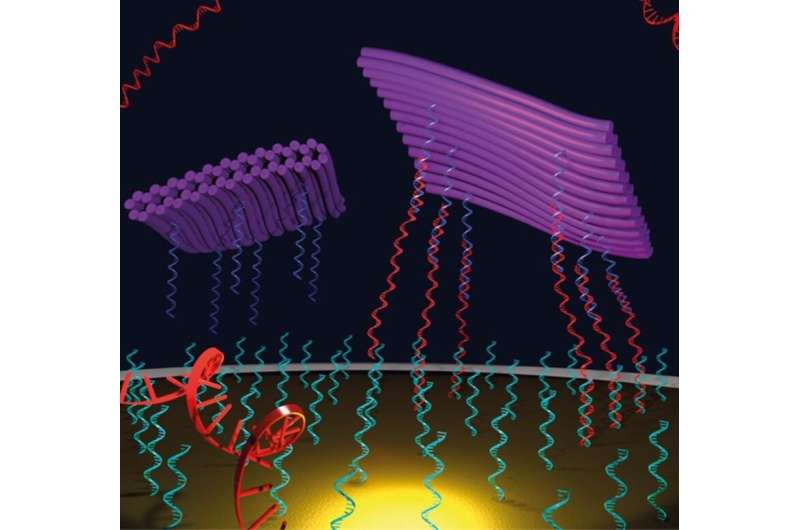
Modulaire en programmeerbare DNA-origamitegels kunnen zich binden aan DNA-sequenties (rood). De resulterende complexen versterken het elektrochemische signaal. Credits: Petteri Piskunen, Universiteit van Aalto
Nieuwe resultaten bieden een platform voor efficiëntere, selectievere en gevoeligere DNA-biosensoren die kunnen worden gebruikt bij het opsporen van verschillende ziekteverwekkers en ziekten.
Elektrochemische DNA-biosensoren zijn veelbelovend voor het monitoren van verschillende ziekten. Over het algemeen zijn hun detectietoepassingen enorm, van doel-DNA-analyten zoals bacteriële genen en tumorsequenties tot bijvoorbeeld klinisch relevante concentraties van SARS CoV-2-biomarkers.
Het is echter een uitdaging om de juiste gevoeligheid en selectiviteit van dergelijke systemen te bereiken en hun vertaling van het laboratorium naar een klinische omgeving mogelijk te maken, aangezien deze benaderingen vaak gepaard gaan met complexe chemie, elektrochemische labeling, technisch uitdagende materialen of meerstapsverwerking.
Nu heeft een team van onderzoekers van Aalto University (Finland) en University of Strathclyde (Glasgow, VK) een manier gevonden om de gevoeligheid van elektrochemische DNA-sensoren aanzienlijk te verbeteren door modulaire DNA-nanostructuren als hun nieuwe componenten te gebruiken. De onderzoekers combineerden de conventionele op DNA gebaseerde sensortechnieken met programmeerbare DNA-origamistructuren om een labelvrije sensor te creëren met een aanzienlijk verhoogde selectiviteit en detectiegevoeligheid.
“In de praktijk is ons uitgangspunt een vrij eenvoudig en algemeen DNA-biosensortype – we hebben een elektrodesysteem ondergedompeld in de analytoplossing, waar de detectie-elektrode is gecoat met enkelstrengige DNA-sondestrengen die complementair zijn aan de (enkelstrengige) ) doel-DNA-sequenties. Zodra de doelstreng bindt en hybridiseert met de sondestreng, bewegen de elektrische ladingen nabij de elektrode een beetje, wat betekent dat we een verandering in het elektrochemische signaal kunnen zien, “legt promovendus Petteri Piskunen van Aalto University, een van de auteurs van het onderzoek.
“Hier spelen DNA-origami-nanostructuren een rol. We hebben onze tegelachtige DNA-origami uitgerust met doelvangende strengen die efficiënt en selectief kunnen binden aan het ene uiteinde van de doelsequentie, terwijl het andere uiteinde van het doel zich bindt aan de sondestrengen. , creëren we een sandwich-achtig complex, waarbij de doelstreng gevangen zit tussen de elektrode en de DNA-origami. In plaats van een kleine signaalverandering op de doelbinding te registreren, zullen we een versterkt effect zien door de aanwezigheid van de relatief grote DNA-origami”, vervolgt Piskunen.
“We hebben de haalbaarheid van ons systeem aangetoond door een genfragment te detecteren van bacteriën met een antibioticaresistentie. We konden dit doelwit selectief vangen vanuit een vrij complexe oplossing die verschillende soorten enkelstrengs DNA bevatte, van korte strengen en junk-fragmenten tot lange circulaire DNA. Met onze sensor konden we op betrouwbare wijze 100-1000 keer lagere doelconcentraties detecteren dan met de conventionele technieken”, zegt Visiting Scientist Veikko Linko (momenteel universitair hoofddocent aan de Universiteit van Tartu, Estland).
“Het is bemoedigend om te denken dat we door veelzijdige DNA-origami te combineren met bijvoorbeeld bedrukbare en wegwerpelektroden, labelvrije detectieplatforms kunnen creëren met zo’n hoge gevoeligheid en specificiteit. Dit positioneert onze technologie met een route naar massaproductie en brede toepasbaarheid als punt Op dit moment wordt er samengewerkt met de Universiteit van Strathclyde om de sensoropstelling te generaliseren voor gebruik met verschillende soorten biomarkers, “concludeert Linko.
De bevindingen worden gepubliceerd in het tijdschrift ACS-sensoren.
Meer informatie:
Paul Williamson et al., Signaalversterking in elektrochemische DNA-biosensoren met behulp van origami-tegels voor het vastleggen van DNA, ACS-sensoren (2023). DOI: 10.1021/acssensors.2c02469
Tijdschrift informatie:
ACS-sensoren
Aangeboden door Aalto Universiteit
