Krediet: Universiteit van Kanazawa
In organismen waarvan de cellen een kern hebben, zoals planten en dieren, zijn de basisverpakkingseenheden van DNA de zogenaamde nucleosomen. Een nucleosoom bestaat uit een stuk DNA dat is gewikkeld rond acht eiwitten die histonen worden genoemd. Genexpressie, die aan de basis ligt van eiwitproductie, vereist het ‘lezen’ van DNA, waarvoor DNA tijdelijk moet worden uitgepakt. Gedetailleerde studies, en vooral visualisaties, van DNA-histon- en nucleosoomdynamiek zijn cruciaal voor een beter begrip van het uitpakken van DNA en gerelateerde processen.
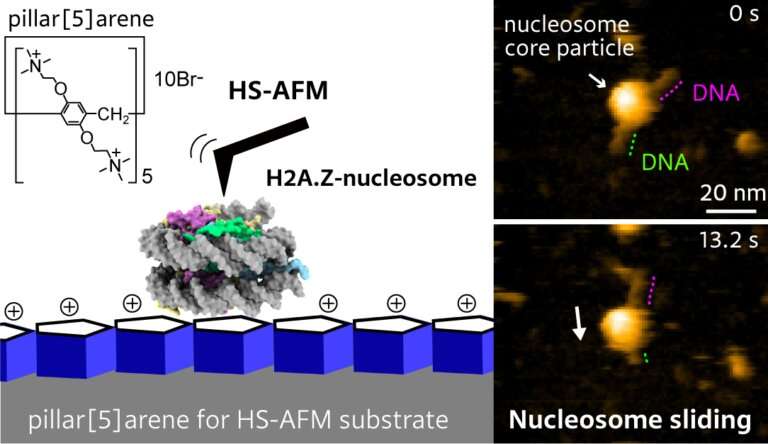
Mikihiro Shibata van Kanazawa University en collega’s zijn er nu in geslaagd video-opnamen te maken van de nucleosoomdynamiek van H2A.Z, een histonvariant die wordt geassocieerd met verschillende biologische processen. De video’s onthullen het spontane glijden van H2A.Z-nucleosomen op een substraat. Het werk wordt gepubliceerd in het tijdschrift Nano-brieven.
Histone-varianten, zoals H2A.Z, verschillen van de canonieke vormen (zoals H2A) die men tegenkomt in stabiele nucleosoomverpakkingen. Ze vormen onstabiele nucleosomen met bepaalde biologische functies; Aangenomen wordt dat H2A.Z een rol speelt bij de vroege embryonale ontwikkeling en stamceldifferentiatie. De dynamiek van het H2A.Z-nucleosoom onder fysiologische omstandigheden is grotendeels onbekend. Shibata en collega’s gebruikten snelle atoomkrachtmicroscopie (HS-AFM) om de dynamiek van H2A.Z-nucleosoom te onderzoeken, omdat de methode een krachtig hulpmiddel voor nano-imaging is voor het visualiseren van moleculaire structuren en hun dynamiek met een hoge spatiotemporele resolutie.
Om de DNA-histondynamiek in HS-AFM-experimenten te observeren, moet het nucleosoom op een substraat worden geplaatst. Het DNA zou gemakkelijk aan het substraat moeten adsorberen, maar tegelijkertijd zouden substraat-DNA-interacties nog steeds zwak genoeg moeten zijn om te voorkomen dat dynamische processen worden onderdrukt. De wetenschappers bereidden daarom substraten voor door pilaar te plaatsen[5]arenes op een mica-oppervlak. De pijler[5]arenes, moleculen met een vijfhoekige buisvormige structuur, vormen een dunne film op het mica en vormen het ideale oppervlak voor observaties van nucleosoomdynamica.
De onderzoekers keken naar de tijdsevolutie van een systeem dat bestaat uit een nucleosoomdeeltje op een DNA-streng. Experimenten met canonieke H2A-histonen bevestigden de stabiliteit van H2A-nucleosomen: er werden geen significante veranderingen in de tijd waargenomen. Waarnemingen voor H2A.Z-histonvarianten lieten echter een ander beeld zien. HS-AFM met een tijdresolutie van 0,3 s onthulde glijdende gebeurtenissen, waarbij een nucleosoomdeeltje langs de DNA-streng glijdt.
De bevindingen van Shibata en collega’s kunnen leiden tot een beter begrip van de biochemische mechanismen achter genexpressie. “De hier gepresenteerde beeldvorming met één molecuul door HS-AFM zou in de nabije toekomst kunnen helpen de relatie tussen nucleosoomdynamiek en genregulatie te onthullen”, zeggen de onderzoekers.
High-speed atoomkrachtmicroscopie
Het algemene principe van atoomkrachtmicroscopie (AFM) is het maken van een zeer kleine puntscan van het oppervlak van een monster. Tijdens deze horizontale (xy) scan volgt de punt, die aan een kleine cantilever is bevestigd, het verticale (z) profiel van het monster, waardoor een kracht op de cantilever wordt opgewekt die kan worden gemeten. De grootte van de kracht op de xy-positie kan worden gerelateerd aan de z-waarde; de xyz-gegevens die tijdens een scan worden gegenereerd, resulteren vervolgens in een hoogtekaart met structurele informatie over het onderzochte monster.
Bij high-speed-AFM (HS-AFM) is het werkingsprincipe iets meer betrokken: de cantilever wordt gemaakt om te oscilleren in de buurt van zijn resonantiefrequentie. Wanneer de tip over een oppervlak wordt bewogen, worden de variaties in de amplitude (of de frequentie) van de oscillatie van de cantilever – als gevolg van de interactie van de tip met het oppervlak van het monster – geregistreerd, aangezien deze een maat vormen voor de lokale “z” -waarde. AFM heeft geen lenzen, dus de resolutie wordt niet beperkt door de zogenaamde diffractielimiet zoals bijvoorbeeld bij röntgendiffractie.
HS-AFM resulteert in een video, waarbij het tijdsinterval tussen de frames afhangt van de snelheid waarmee een enkel beeld kan worden gegenereerd (door het sample xy-scannen). Onderzoekers van Kanazawa University hebben de afgelopen jaren HS-AFM verder ontwikkeld, zodat het kan worden toegepast om biochemische moleculen en biomoleculaire processen real-time te bestuderen. Mikihiro Shibata en collega’s hebben de methode nu toegepast om de nucleosoomdynamiek te bestuderen, waarbij ze een glijdend proces van nucleosoomdeeltjes langs een DNA-streng onthullen.
Meer informatie:
Shin Morioka et al, High-Speed Atomic Force Microscopy onthult spontane nucleosoomverschuiving van H2A.Z op de subseconde tijdschaal, Nano-brieven (2023). DOI: 10.1021/acs.nanolett.2c04346
Tijdschrift informatie:
Nano-brieven
Aangeboden door de Kanazawa-universiteit