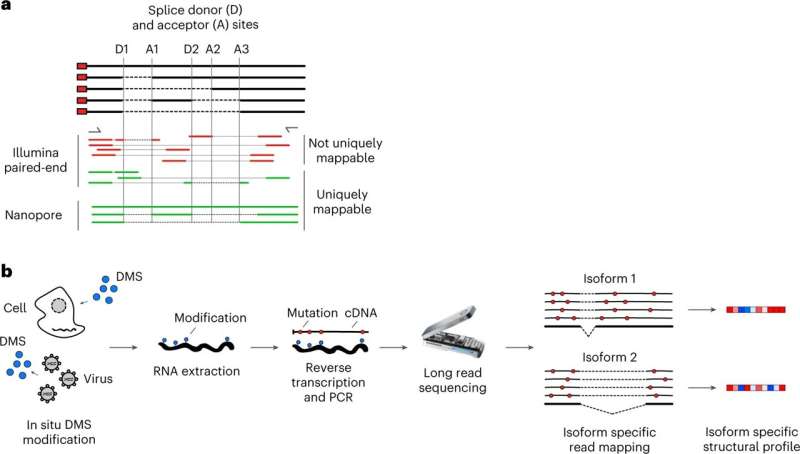
Transcript isovormen structurele analyse door Nano-DMS-MaP. Nano-DMS-MaP voor isovorm-opgeloste RNA-structuurbepaling. A, Transcript-isovormen kunnen worden gegenereerd door alternatieve splitsing. Gemeenschappelijke regio’s tussen isovormen van transcripten kunnen verschillende structuren hebben, maar korte sequencing-lezingen kunnen niet altijd ondubbelzinnig worden toegewezen aan isovormen van transcripten. Lang gelezen nanopore-sequencing kan op unieke wijze worden toegewezen aan isovormen van transcripten. B, kan DMS de RNA-structuur in situ in cellen of virionen onderzoeken. Gemodificeerd RNA wordt geëxtraheerd en omgekeerd getranscribeerd in lange cDNA-moleculen voor sequentiebepaling op nanopore-apparaten. Isoform-specifieke read mapping maakt isoform-specifieke structurele profilering mogelijk. MinION apparaat afbeelding tegoed: Oxford Nanopore Technologies (2023). Credit: Natuur Methoden (2023). DOI: 10.1038/s41592-023-01862-7
Ribonucleïnezuur (RNA) vouwt zich op tot complexe structuren, waardoor het specifiek kan interageren met andere moleculen in de cel. Bij HIV-1 kunnen minieme verschillen in RNA-vouwing cruciaal zijn om te bepalen of viraal RNA “verpakt” is en dus leidt tot virale replicatie.
Dit is nu ontdekt door onderzoekers van het Helmholtz Instituut Würzburg door een methode te verbeteren die wordt gebruikt om de RNA-structuur te bestuderen met een nieuwe sequencing-technologie. Hun bevindingen kunnen helpen bij het ontwerpen van nieuwe antivirale middelen en werden vandaag gepubliceerd in het tijdschrift Natuur Methoden.
Human Immunodeficiency Virussen (HIV) zijn wereldwijd verantwoordelijk voor miljoenen infecties. Door het Acquired Immunodeficiency Syndrome (AIDS) te veroorzaken, hebben deze ziekteverwekkers geleid tot bijna 40 miljoen doden sinds het uitbreken van de hiv-pandemie in de jaren tachtig. Decennia lang hebben wetenschappers onderzoek gedaan naar mogelijke antivirale therapieën en er zijn nu effectieve medicijnen beschikbaar voor geïnfecteerden. Het is echter de combinatie van meerdere antivirale middelen met verschillende doelen die een revolutie teweeg heeft gebracht in de hiv-therapie, en er zijn voortdurend nieuwe medicijnen nodig om de resistentie tegen medicijnen te bestrijden.
“In onze studie introduceren we een mogelijk nieuw doelwit om HIV en andere potentieel zoönotische retrovirussen een stap voor te blijven”, zegt Redmond Smyth. Smyth leidt een onderzoeksgroep aan het Helmholtz Institute for RNA-based Infection Research (HIRI) in Würzburg, een locatie van het Braunschweig Helmholtz Center for Infection Research (HZI) in samenwerking met de Julius-Maximilians-Universität (JMU) Würzburg, en heeft leidde het huidige onderzoek.
“Onze nieuwe methode kan de structurele variaties onderscheiden tussen zeer vergelijkbare RNA’s, zelfs die gecreëerd door splitsing”, legt Patrick Bohn, een Ph.D. student in het Smyth-lab. De onderzoeker is de co-eerste auteur van de studie, die vandaag in het tijdschrift is gepubliceerd Natuur Methoden.
Splicing is een biologisch proces dat in zekere zin de genetische blauwdruk van een cel in het oorspronkelijke boodschapper-RNA verfijnt voor latere vertaling in nieuwe eiwitten. “In hogere organismen genereert splicing eiwitdiversiteit, maar onze bevindingen geven aan dat het ook kan bijdragen aan de biologische functie door nieuwe RNA-structuren te produceren”, zegt Bohn.
Heel vergelijkbaar en toch anders
De huidige bevindingen zijn verkregen door een technologie te verbeteren om te meten hoe RNA in de cel wordt gevouwen. Veel wetenschappers hebben geprobeerd structuren van gesplitst en niet-gesplitst HIV-1-RNA te bestuderen, maar dit was een uitdaging omdat eerdere technologieën de RNA-structuur alleen in kleine fragmenten maten. Wetenschappers van het HIRI hebben nu long-read sequencing toegepast om de RNA-structuur over de volledige lengte van het RNA-molecuul te bestuderen, en gebruikten het om te laten zien hoe het HIV-1-virus zijn RNA van volledige lengte selecteert om het in virale deeltjes te verpakken.
“We hebben onthuld dat hiv-1-RNA heel anders vouwt wanneer het wordt gesplitst – een ontdekking die het belang onthult van het bestuderen van biologische processen, rekening houdend met de complexe oorspronkelijke omgeving”, zegt Anne-Sophie Gribling-Burrer, een postdoctoraal onderzoeker en co -eerste auteur op het papier. Als gevolg hiervan bleek dat gesplitste versies van HIV-1-RNA niet verpakt waren.
“Gesplitste RNA’s hebben niet enkele van de structurele kenmerken die nodig zijn om verpakt te worden, waardoor ze een mechanisme bieden voor verpakkingsselectiviteit en de replicatie van het virus beïnvloeden”, legt Gribling-Burrer uit.
“Het begrijpen van dit mechanisme is een belangrijke stap in het ontwikkelen van nieuwe antivirale middelen tegen een breed scala aan retrovirussen”, stelt Redmond Smyth. Daarnaast denken de onderzoekers dat hun methode in de toekomst nuttig zal zijn voor een breed veld van moleculair biologen op zeer verschillende gebieden.
Technische achtergrond
Enkelstrengs RNA kan door basenparing tot complexe structuren worden gevouwen. Genoombrede metingen van de RNA-structuur kunnen worden verkregen met behulp van reagentia die reageren met ongepaarde basen, wat leidt tot adducten die kunnen worden geïdentificeerd door mutatieprofilering op sequencing-machines van de volgende generatie.
Een nadeel van deze experimenten is dat korte sequencing-lezingen zelden kunnen worden toegewezen aan specifieke transcriptie-isovormen. Bijgevolg wordt informatie verkregen als een populatiegemiddelde in regio’s die worden gedeeld door transcripties, waardoor het onderliggende structurele landschap vervaagt.
In hun studie introduceren wetenschappers van het Helmholtz Instituut Würzburg nanopore dimethyl-sulfaat mutatieprofilering (Nano-DMS-MaP), een methode die isovorm-opgeloste structurele informatie verschaft van zeer vergelijkbare RNA-moleculen.
Meer informatie:
Patrick Bohn et al, Nano-DMS-MaP maakt isovormspecifieke RNA-structuurbepaling mogelijk, Natuur Methoden (2023). DOI: 10.1038/s41592-023-01862-7
Tijdschrift informatie:
Natuur Methoden
Aangeboden door Helmholtz Vereniging van Duitse onderzoekscentra