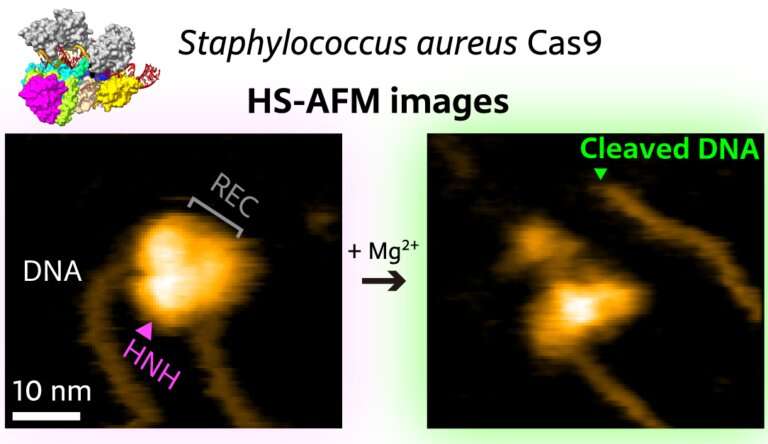
High-speed atomic force microscopie visualisatie van het knippen van een stukje DNA door SaCas9. Krediet: Puppulin et al, ACS Nano
Dat melden onderzoekers van Kanazawa University ACS Nano hoe supersnelle atoomkrachtmicroscopie kan worden gebruikt om de biomoleculaire mechanismen die ten grondslag liggen aan genbewerking te bestuderen.
Het is bekend dat het DNA van prokaryoten – eencellige organismen, bijvoorbeeld bacteriën – sequenties bevat die zijn afgeleid van DNA-fragmenten van virussen die de prokaryoot eerder hebben geïnfecteerd. Deze sequenties, gezamenlijk aangeduid als CRISPR, voor “geclusterde, regelmatig tussen elkaar geplaatste korte palindroomherhalingen”, spelen een belangrijke rol in het antivirale afweersysteem van bacteriën, omdat ze de herkenning en daaropvolgende neutralisatie van infecterende virussen mogelijk maken. Dit laatste wordt gedaan door het enzym Cas9 (“CRISPR-associated protein 9”), een biomolecuul dat lokaal DNA kan afwikkelen, controleren op het bestaan van de CRISPR-sequentie en, indien gevonden, het DNA knippen.
In de afgelopen jaren is CRISPR/Cas9 naar voren gekomen als een hulpmiddel voor het bewerken van het genoom, gebaseerd op het idee dat het Cas9-eiwit kan worden geactiveerd met kunstmatig gecreëerde CRISPR-achtige sequenties. Soms wordt echter het verkeerde doelwit “gevangen” door Cas9 – wanneer de verkeerd geïdentificeerde DNA-sequentie te veel lijkt op de beoogde doelwitsequentie. Het is daarom van cruciaal belang om volledig te begrijpen hoe Cas9 DNA bindt, “ondervraagt” en knipt. Mikihiro Shibata van Kanazawa University en collega’s zijn er nu in geslaagd om de DNA-binding en splijtdynamiek van Staphylococcus aureus (een bepaalde bacterie) Cas9 op video vast te leggen door middel van snelle atoomkrachtmicroscopie (HS-AFM). Hun observaties zullen helpen om een vollediger begrip van CRISPR-Cas9-mechanismen te krijgen.
In de afgelopen jaren is HS-AFM naar voren gekomen als een krachtig hulpmiddel voor nanoimaging voor het bestuderen van moleculaire structuren en hun dynamiek met een hoge spatiotemporele resolutie. Om dergelijke moleculaire dynamiek waarneembaar te maken, moeten monsters op een zorgvuldig gekozen substraat worden geplaatst. Voor hun studie van Staphylococcus aureus Cas9 (SaCas9) hebben Shibata en collega’s een mica-oppervlak chemisch gemodificeerd. Op deze manier “kleven” waargenomen moleculen aan het substraat, maar niet te sterk, zodat de moleculaire mobiliteit nog steeds hoog genoeg is om de relevante biomoleculaire interacties te laten plaatsvinden – vertraagd, binnen tijdsintervallen die toegankelijk zijn voor HS-AFM.
De kunstmatige activering van Cas9 gebeurt via de associatie met een zogenaamd single-guide RNA (sgRNA) molecuul, dat de informatie geeft over de beoogde DNA-sequentie. De wetenschappers merkten eerst op dat het SaCas9-sgRNA-complex een flexibele modulaire structuur aanneemt die kan overschakelen van een open naar een gesloten configuratie, wat uiteindelijk binding aan het DNA mogelijk maakt. Ze slaagden er ook in om het knippen van DNA op de beoogde plek in beeld te brengen.
De onderzoekers keken in meer detail naar het mechanisme van detectie van gericht DNA door SaCas9-sgRNA en vonden bewijs dat het om een selectieve interactie op lange afstand gaat. Hoewel de precieze aard van deze interactie onduidelijk blijft, geloven Shibata en collega’s dat hydrofobe krachten tussen delen van het SaCas9-sgRNA-complex en het DNA een sleutelrol spelen.
Het potentiële belang van dergelijke langeafstandsinteracties bij CRISPR-Cas9-genoombewerking is onverwacht – tot nu toe was de algemene overtuiging dat doel-DNA wordt geïdentificeerd door middel van diffusieprocessen. Aanvullende studies zijn nodig om dit aspect verder te onderzoeken. De wetenschappers stellen: “Hoewel de huidige HS-AFM-waarnemingen direct bewijs vormen van de selectieve interactie over lange afstand tussen SaCas9-sgRNA en zijn doelwit-DNA, zijn verder onderzoek nodig om het mechanisme van het zoeken naar doelwit-DNA te bevestigen, zoals verondersteld en geïllustreerd in de huidige studie.”
High-speed atoomkrachtmicroscopie
Het algemene principe van atoomkrachtmicroscopie (AFM) is om met een zeer kleine tip het oppervlak van een monster te scannen. Tijdens deze horizontale (xy) scan volgt de punt, die aan een kleine cantilever is bevestigd, het verticale (z) profiel van het monster, waardoor een kracht op de cantilever wordt opgewekt die kan worden gemeten. De grootte van de kracht op de xy-positie kan worden gerelateerd aan de z-waarde; de xyz-gegevens die tijdens een scan worden gegenereerd, resulteren vervolgens in een hoogtekaart met structurele informatie over het onderzochte monster.
Bij high-speed-AFM (HS-AFM) is het werkingsprincipe iets meer betrokken: de cantilever wordt gemaakt om te oscilleren in de buurt van zijn resonantiefrequentie. Wanneer de tip over een oppervlak wordt bewogen, worden de variaties in de amplitude (of de frequentie) van de oscillatie van de cantilever – die het resultaat is van de interactie van de tip met het oppervlak van het monster – geregistreerd, aangezien deze een maat vormen voor de lokale “z” -waarde. AFM heeft geen lenzen, dus de resolutie wordt niet beperkt door de zogenaamde diffractielimiet zoals bijvoorbeeld bij röntgendiffractie.
HS-AFM resulteert in een video, waarbij het tijdsinterval tussen de frames afhangt van de snelheid waarmee een enkel beeld kan worden gegenereerd (door het sample xy-scannen). Onderzoekers van Kanazawa University hebben de afgelopen jaren HS-AFM verder ontwikkeld, zodat het kan worden toegepast om biochemische moleculen en biomoleculaire processen real-time te bestuderen. Mikihiro Shibata en collega’s hebben de methode nu toegepast om de moleculaire dynamiek van een Cas9-DNA-interactieproces te bestuderen, wat zeer relevant is voor lopend onderzoek naar de CRISPR-Cas9-tool voor genoombewerking.
Meer informatie:
Leonardo Puppulin et al, Dynamics of Target DNA Binding and Cleavage by Staphylococcus aureus Cas9 zoals onthuld door High-Speed Atomic Force Microscopy, ACS Nano (2023). DOI: 10.1021/acsnano.2c10709
Tijdschrift informatie:
ACS Nano
Aangeboden door de Kanazawa-universiteit