Snelle visualisatie van atoomkrachtmicroscopie van de nucleosoomdynamiek met canonieke (bovenste) en staartloze (onderste) histonen. Credit: Nano-brieven (2024). DOI: 10.1021/acs.nanolett.4c00801
Bij planten en dieren zijn de basisverpakkingseenheden van DNA, die genetische informatie bevatten, de zogenaamde nucleosomen. Een nucleosoom bestaat uit een stukje DNA dat rond acht eiwitten is gewikkeld die bekend staan als histonen.
Tijdens genexpressie (het proces dat aan de basis ligt van de eiwitproductie) zijn nucleosomen betrokken bij verschillende dynamische structurele veranderingen, zoals het verschuiven van het nucleosoom, het uitpakken van DNA en andere DNA-histone-interacties. Van bijzonder belang bij deze processen zijn de eindstructuren, of staarten, van de histonen.
Histone-staarten ondergaan chemische modificaties, waardoor de functionaliteit van de histon indien nodig verandert. Gedetailleerde studies, en vooral visualisaties, van de nucleosoomdynamiek zijn cruciaal voor een beter begrip van de rol van histonstaarten.
Mikihiro Shibata van de Kanazawa Universiteit en collega's zijn er nu in geslaagd video-opnames te maken van staartloze nucleosomen, wat aantoont dat de afwezigheid van histonstaarten de dynamische activiteit van een nucleosoom aanzienlijk verhoogt. De studie is gepubliceerd in Nano-brieven.
De wetenschappers gebruikten snelle atomaire krachtmicroscopie (HS-AFM), een krachtig hulpmiddel voor nano-imaging voor het visualiseren van moleculaire structuren en hun dynamiek met een hoge ruimtelijke en temporele resolutie.
Hiervoor moesten de nucleosomen op een substraat worden geplaatst. Shibata en collega's gebruikten een filmpje van zogenaamde pilaren[5]arenes (moleculen met een vijfhoekige buisvormige structuur) als substraat en vormen een ideaal oppervlak omdat de nucleosomen er gemakkelijk aan worden geadsorbeerd zonder dat dynamische processen worden onderdrukt.
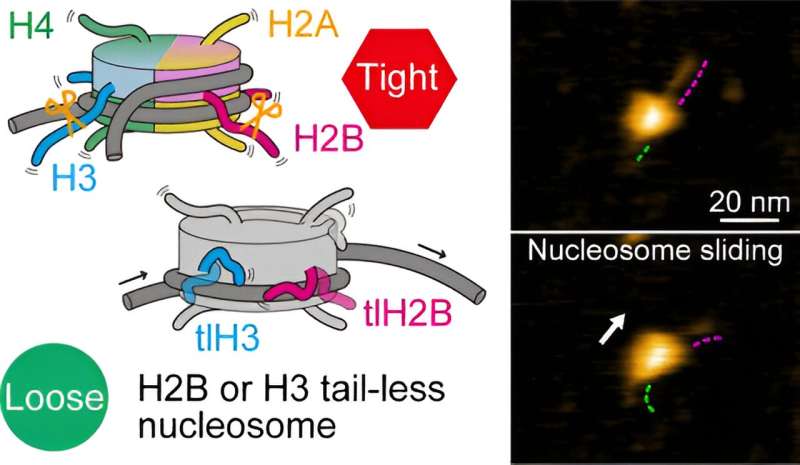
De onderzoekers keken eerst naar nucleosomen waarvan alle acht histonen geen staart hadden. Op basis van hun HS-AFM-waarnemingen concludeerden ze dat het verschuiven van nucleosomen en het uitpakken/opnieuw inpakken van DNA vaker voorkwamen dan bij normale (canonieke) nucleosomen. Dit suggereert dat zonder staarten de histon-DNA-interactie verzwakt is, wat leidt tot een situatie waarin DNA gemakkelijker kan loskomen van de histonen.
Om de rol van specifieke histonstaarten beter te begrijpen, hebben Shibata en collega's nucleosomen voorbereid waarbij één type histon staartloos was. Er zijn vier verschillende soorten histonen, genaamd H2A, H2B, H3 en H4. HS-AFM-experimenten met de nucleosomen onthulden dat H2B- en H3-staartloze nucleosomen een verhoogde frequentie van dynamiek vertoonden. Omgekeerd betekent dit dat canonieke H2B- en H3-histonen essentieel zijn voor de stabiliteit van het nucleosoom.
De wetenschappers wijzen erop dat ze geen enkele daadwerkelijke beweging van histonstaarten konden waarnemen – hoogstwaarschijnlijk was de temporele resolutie van het onderzoek, 0,3 seconden, veel langzamer dan de snelheid van de dynamiek van het in- en uitpakken van de staarten. Ondanks deze beperking bewijst het werk van Shibata en collega's duidelijk dat de staarten van H2B- en H3-histonen de belangrijkste bijdrage leveren aan de nucleosoomdynamiek.
Met betrekking tot toekomstig werk, aldus de onderzoekers: “een techniek voor het labelen van histonstaartpunten zou HS-AFM in staat kunnen stellen de bewegingen van de histonstaarten zelf vast te leggen.”
Hogesnelheidsmicroscopie met atoomkracht
Het algemene principe van atomaire krachtmicroscopie (AFM) is om met een zeer kleine punt het oppervlak van een monster te scannen. Tijdens deze horizontale (xy) scan volgt de punt, die aan een kleine cantilever is bevestigd, het verticale (z) profiel van het monster, waardoor een kracht op de cantilever wordt uitgeoefend die kan worden gemeten.
De grootte van de kracht op de xy-positie kan gerelateerd zijn aan de z-waarde; de tijdens een scan gegenereerde xyz-gegevens resulteren vervolgens in een hoogtekaart die structurele informatie over het onderzochte monster biedt. Bij hogesnelheids-AFM (HS-AFM) is het werkingsprincipe iets ingewikkelder: de cantilever is gemaakt om te oscilleren nabij zijn resonantiefrequentie.
Wanneer de punt over een oppervlak wordt bewogen, worden de variaties in de amplitude (of de frequentie) van de oscillatie van de cantilever (die het gevolg zijn van de interactie van de punt met het oppervlak van het monster) geregistreerd, aangezien deze een maatstaf vormen voor de lokale z-waarde. Bij AFM zijn geen lenzen nodig, dus de resolutie wordt niet beperkt door de zogenaamde diffractielimiet zoals bijvoorbeeld bij röntgendiffractie.
HS-AFM resulteert in een video, waarbij het tijdsinterval tussen frames afhangt van de snelheid waarmee een enkel beeld kan worden gegenereerd (door het monster xy-scannen).
Onderzoekers van het Nano Life Science Institute (WPI-NanoLSI), Kanazawa University, hebben HS-AFM de afgelopen jaren verder ontwikkeld, zodat het kan worden toegepast om biochemische moleculen en biomoleculaire processen in realtime te bestuderen. Mikihiro Shibata en collega's hebben de methode nu toegepast om de nucleosoomdynamiek in detail te bestuderen, en in het bijzonder de rol van de moleculaire uiteinden van histonen – eiwitten die een cruciale rol spelen bij de toegankelijkheid van DNA.
Meer informatie:
Shin Morioka et al., High-Speed Atomic Force Microscopie onthult de nucleosoomverschuiving en de DNA-uitpak-/inpakdynamiek van staartloze nucleosomen, Nano-brieven (2024). DOI: 10.1021/acs.nanolett.4c00801
Tijdschriftinformatie:
Nano-brieven
Geleverd door de Kanazawa Universiteit