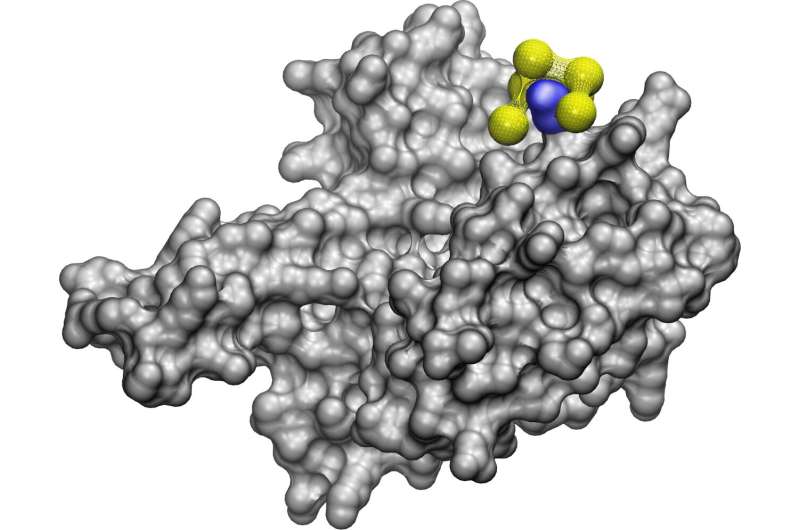
Het nieuwe computermodel, NeCLAS, voorspelt dat een nanodeeltje, weergegeven als een set gele ballen die zijn vastgemaakt door netten, netjes past rond een heel specifiek uitsteeksel op een eiwit, gemarkeerd in blauw. De bindingsplaats wordt door experimenten bevestigd. Dit soort nanodeeltjes, moleculaire pincetten genoemd, kunnen worden gebruikt om de werking van ziekteverwekkers en toxische eiwitophopingen in de war te sturen. Credits: Paolo Elvati, Violi Lab, Universiteit van Michigan
Identificeren of en hoe een nanodeeltje en een eiwit zich aan elkaar zullen binden, is een belangrijke stap in de richting van het kunnen ontwerpen van antibiotica en antivirale middelen op aanvraag, en een computermodel dat is ontwikkeld aan de Universiteit van Michigan kan dit doen.
De nieuwe tool kan helpen bij het vinden van manieren om antibioticaresistente infecties en nieuwe virussen te stoppen, en kan helpen bij het ontwerpen van nanodeeltjes voor verschillende doeleinden.
“Alleen al in 2019 bedroeg het aantal mensen dat stierf aan antimicrobiële resistentie 4,95 miljoen. Zelfs vóór COVID, dat het probleem verergerde, toonden studies aan dat tegen 2050 het aantal sterfgevallen door antibioticaresistentie 10 miljoen zal zijn”, zei Angela Violi, een Arthur F. Thurnau hoogleraar werktuigbouwkunde, en overeenkomstige auteur van de studie die de omslag maakte van Natuur Computational Science.
“In mijn ideale scenario, over 20 of 30 jaar, zou ik graag – gegeven een superbug – in staat zijn om snel de beste nanodeeltjes te produceren die het kunnen behandelen.”
Veel van het werk in cellen wordt gedaan door eiwitten. Interactieplaatsen op hun oppervlak kunnen moleculen aan elkaar hechten, uit elkaar halen en andere modificaties uitvoeren: doorgangen naar cellen openen, suikers afbreken om energie vrij te maken, structuren bouwen om groepen cellen te ondersteunen en meer. Als we medicijnen zouden kunnen ontwerpen die zich richten op cruciale eiwitten in bacteriën en virussen zonder onze eigen cellen te beschadigen, zou dat de mens in staat stellen snel nieuwe en veranderende ziekten te bestrijden.
Het nieuwe model, genaamd NeCLAS, maakt gebruik van machine learning, de AI-techniek die de virtuele assistent op uw smartphone en ChatGPT aandrijft. Maar in plaats van taal te leren verwerken, absorbeert het structurele modellen van eiwitten en hun bekende interactieplaatsen. Uit deze informatie leert het om te extrapoleren hoe eiwitten en nanodeeltjes kunnen interageren, om bindingsplaatsen en de waarschijnlijkheid van binding daartussen te voorspellen, en om interacties tussen twee eiwitten of twee nanodeeltjes te voorspellen.
“Er bestaan andere modellen, maar de onze is het beste voor het voorspellen van interacties tussen eiwitten en nanodeeltjes”, zegt Paolo Elvati, UM-onderzoeker in werktuigbouwkunde.
AlphaFold is bijvoorbeeld een veelgebruikt hulpmiddel om de 3D-structuur van een eiwit te voorspellen op basis van zijn bouwstenen, de zogenaamde aminozuren. Hoewel deze capaciteit cruciaal is, is dit nog maar het begin: ontdekken hoe deze eiwitten zich assembleren tot grotere structuren en het ontwerpen van praktische systemen op nanoschaal zijn de volgende stappen.
“Dat is waar NeCLAS om de hoek komt kijken”, zegt Jacob Saldinger, UM-promovendus chemische technologie en eerste auteur van het onderzoek. “Het gaat verder dan AlphaFold door te laten zien hoe nanostructuren met elkaar omgaan, en het is niet beperkt tot eiwitten. Hierdoor kunnen onderzoekers de potentiële toepassingen van nanodeeltjes begrijpen en hun ontwerpen optimaliseren.”
Het team testte drie casestudy’s waarvoor ze aanvullende gegevens hadden:
- Moleculair pincet, waarbij een molecuul zich bindt aan een bepaalde plek op een ander molecuul. Deze aanpak kan schadelijke biologische processen stoppen, zoals de opeenhoping van eiwitplaques bij hersenziekten zoals de ziekte van Alzheimer.
- Hoe kwantumstippen van grafeen de biofilm breken die door stafylokokbacteriën wordt geproduceerd. Deze nanodeeltjes zijn koolstofschilfers, niet meer dan enkele atoomlagen dik en 0,0001 millimeter uit elkaar. Het doorbreken van biofilms is waarschijnlijk een cruciaal hulpmiddel bij het bestrijden van antibioticaresistente infecties, waaronder de superbacterie methicilline-resistente Staphylococcus aureus (MRSA), die gewoonlijk in ziekenhuizen wordt verkregen.
- Of kwantumdots van grafeen zich in water zouden verspreiden, wat aantoont dat het model in staat is om de binding van nanodeeltjes en nanodeeltjes te voorspellen, ook al was het uitsluitend getraind op eiwit-eiwitgegevens.
Hoewel veel eiwit-eiwitmodellen aminozuren instellen als de kleinste eenheid waarmee het model rekening moet houden, werkt dit niet voor nanodeeltjes. In plaats daarvan stelde het team de grootte van dat kleinste kenmerk in op ongeveer de grootte van het aminozuur, maar liet het computermodel vervolgens beslissen waar de grenzen tussen deze minimale kenmerken lagen. Het resultaat zijn representaties van eiwitten en nanodeeltjes die een beetje lijken op verzamelingen onderling verbonden kralen, wat meer flexibiliteit biedt bij het verkennen van kleinschalige interacties.
“Behalve dat NeCLAS algemener is, gebruikt het ook veel minder trainingsgegevens dan AlphaFold. We hebben maar 21 nanodeeltjes om naar te kijken, dus we moeten eiwitgegevens op een slimme manier gebruiken”, zegt Matt Raymond, UM-promovendus in elektrotechniek en computertechniek. en studie co-auteur.
Vervolgens is het team van plan andere biofilms en micro-organismen, waaronder virussen, te onderzoeken.
Meer informatie:
Jacob Charles Saldinger et al, Domein-agnostische voorspellingen van interacties op nanoschaal in eiwitten en nanodeeltjes, Natuur Computational Science (2023). DOI: 10.1038/s43588-023-00438-x
Tijdschrift informatie:
Natuur Computational Science
Aangeboden door de Universiteit van Michigan