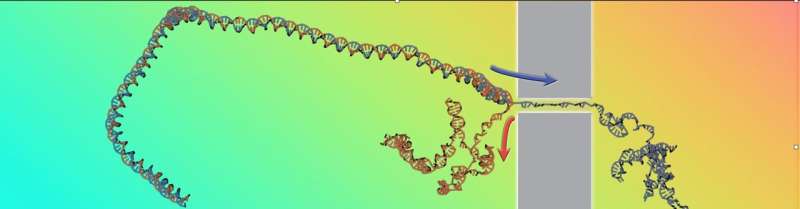
Het onderzoeksteam gebruikte een cluster van computers om de translocatie van DNA door nanoporiën te simuleren met verschillende drijvende krachten die de uitpaksnelheid van het DNA bijhielden, een type gegevens dat zelden is bestudeerd, ondanks dat ze direct toegankelijk waren in experimenten. ”, met behulp van de informatie over de snelheid om de thermodynamica van de vorming en breuk van de dubbele helixstructuur nauwkeurig te reconstrueren. Credits: Antonio Suma en Cristian Micheletti
Nauwkeurig reconstrueren hoe de delen van een complexe molecuul bij elkaar worden gehouden en alleen weten hoe het molecuul vervormt en uiteenvalt – dit was de uitdaging die een onderzoeksteam onder leiding van SISSA’s Cristian Micheletti aanging en onlangs gepubliceerd op Fysieke beoordelingsbrieven. In het bijzonder bestudeerden de wetenschappers hoe een dubbele DNA-helix openritst wanneer deze met hoge snelheid door een nanogaatje wordt verplaatst, waarbij fundamentele thermodynamische eigenschappen van DNA worden gereconstrueerd op basis van de enige snelheid van het proces.
De translocatie van polymeren door nanoporiën is lang bestudeerd als een fundamenteel theoretisch probleem en ook vanwege de verschillende praktische vertakkingen ervan, bijvoorbeeld voor genoomsequencing. We herinneren ons dat dit laatste inhoudt dat een DNA-filament door een porie wordt gedreven die zo smal is dat slechts één van de dubbele helixstrengen kan passeren, terwijl de andere streng achterblijft. Als gevolg hiervan zal de getransloceerde dubbele DNA-helix noodzakelijkerwijs splitsen en ontspannen, een effect dat bekend staat als uitpakken.
Het onderzoeksteam, waartoe ook Antonio Suma van de Universiteit van Bari, eerste auteur, en Vincenzo Carnevale van Temple University behoren, gebruikte een cluster van computers om het proces te simuleren met verschillende drijvende krachten die de uitpaksnelheid van het DNA bijhielden, een soort gegevens dat zelden is bestudeerd, ondanks dat het direct toegankelijk is in experimenten.
Met behulp van eerder ontwikkelde theoretische en wiskundige modellen konden onderzoekers “achteruit” werken, waarbij ze de informatie over de snelheid gebruikten om de thermodynamica van de vorming en breuk van de dubbele helixstructuur nauwkeurig te reconstrueren.
“Eerdere theorieën”, leggen de onderzoekers uit, “gingen uit van gedetailleerde kennis van de thermodynamica van een moleculair systeem dat vervolgens werd gebruikt om de reactie op min of meer invasieve externe spanningen te voorspellen. Dit alleen al is een grote uitdaging op zich. We hebben gekeken naar het omgekeerde probleem: we zijn uitgegaan van de reactie van het DNA op agressieve spanningen, zoals het geforceerd openritsen van de dubbele helix, om de details van de thermodynamica te herstellen.”
“Vanwege de invasieve en snelle aard van het uitpakproces leek het project gedoemd te mislukken, en dat was waarschijnlijk de reden waarom het nog nooit eerder was geprobeerd. We wisten echter ook dat de juiste theoretische en wiskundige modellen, indien van toepassing, ons een veelbelovende oplossing voor het probleem. Na analyse van de uitgebreide set verzamelde gegevens waren we zeer verheugd om te ontdekken dat dit precies het geval was; we waren blij dat we de juiste intuïtie hadden.”
De techniek die in het onderzoek is gebruikt, is algemeen en daarom verwachten de onderzoekers dat ze deze kunnen uitbreiden buiten DNA naar andere moleculaire systemen die nog relatief onontgonnen zijn. Een goed voorbeeld zijn de zogenaamde moleculaire motoren, eiwitaggregaten die energie gebruiken om cyclische transformaties door te voeren, vergelijkbaar met de motoren in ons dagelijks leven.
“Tot nu toe”, benadrukken de onderzoekers, “zijn studies over moleculaire motoren begonnen met het formuleren van hypothesen over hun thermodynamica en vervolgens het vergelijken van voorspellingen met experimentele gegevens. evenwichtsexperimenten om de thermodynamica te herstellen, met duidelijke conceptuele en praktische voordelen.”
Meer informatie:
Antonio Suma et al, Onequilibrium Thermodynamics of DNA Nanopore Unzipping, Fysieke beoordelingsbrieven (2023). DOI: 10.1103/PhysRevLett.130.048101
Tijdschrift informatie:
Fysieke beoordelingsbrieven
Aangeboden door International School of Advanced Studies (SISSA)