Grafisch abstract . Credit: ACS Nano (2024). DOI: 10.1021/acsnano.3c10575
Moleculaire computercomponenten kunnen een nieuwe IT-revolutie betekenen en ons helpen goedkopere, snellere, kleinere en krachtigere computers te maken. Toch hebben onderzoekers moeite om manieren te vinden om ze betrouwbaarder en efficiënter samen te stellen.
Om dit te helpen bereiken, onderzochten wetenschappers van het Instituut voor Natuurkunde van de Tsjechische Academie van Wetenschappen de mogelijkheden van zelfassemblage van moleculaire machines, voortbouwend op oplossingen die zijn aangescherpt door natuurlijke evolutie en gebruikmakend van synergie met de huidige chipproductie.
Er is een grens aan de miniaturisatie van de huidige op silicium gebaseerde computerchips. Moleculaire elektronica, die gebruik maakt van schakelaars en geheugens ter grootte van één molecuul, zou een revolutie teweeg kunnen brengen in de omvang, snelheid en mogelijkheden van computers en tegelijkertijd het toenemende stroomverbruik kunnen verminderen, maar hun massaproductie is een uitdaging. Grootschalige, laag-defecte, toegankelijke nanofabricage en assemblage van de componenten blijft ongrijpbaar. Inspiratie uit de levende natuur zou deze status quo kunnen veranderen.
Kleine prototypes van moleculaire circuits die uit een paar moleculen bestaan, worden momenteel geproduceerd door middel van scanning-sondemicroscopie, waarbij ze molecuul per molecuul worden gemanipuleerd door een langzame, zware macroscopische cantilever.
Prokop Hapala, die leiding gaf aan de studie gepubliceerd in ACS Nano, vergelijkt het met het bouwen van een delicaat mozaïek met behulp van een enorme kraan, tegel voor tegel. Zelfassemblage zou dit probleem kunnen oplossen, maar het brengt ook andere uitdagingen met zich mee. Hoe produceren we bijvoorbeeld een verscheidenheid aan structuren als slechts een kleine hoeveelheid structurele informatie kan worden gecodeerd in interacties tussen een paar functionele groepen?
Onderzoekers van het Instituut voor Natuurkunde van de Tsjechische Academie van Wetenschappen haalden inspiratie uit de natuur, waar functionele en structurele componenten zijn ontkoppeld in polymeersjablonen zoals DNA of RNA. Daar vertegenwoordigen suikerfosfaten de basis en zorgen de nucleobasen, gebonden door waterstofbruggen, voor de informatieopslag.
Dankzij deze bindingen kunnen deze informatiepolymeren zichzelf assembleren tot complexe vormen en zelfreplicatie of synthese van andere, kleinere moleculen aandrijven. Deze aanpak is al toegepast in ‘DNA-origami’, waarmee complexe moleculen met de gewenste vormen en functies kunnen worden geproduceerd. Maar hoe kunnen we het proces opschalen en meer variatie bereiken?
“Bekende DNA-basenparen – waarvan je naïef kunt denken dat ze de beste keuze zouden zijn – kunnen niet worden gebruikt zoals ze zijn”, legt Paolo Nicolini, een van de auteurs, uit. “Ze werken prima in de cel, maar dit komt door de omgeving en de rest van de cellulaire machinerie. In omstandigheden die compatibel zijn met nanofabricage zijn ze simpelweg niet selectief genoeg.”
Mithun Manikandan, Paolo Nicolini en Prokop Hapala besloten de mogelijkheden van DNA-origami en fotolithografie te combineren om complexe structuren van hedendaagse chips in kaart te brengen. Dit zou de weg kunnen vrijmaken voor massaproductie van revolutionaire moleculaire circuits, geïntegreerd met de hedendaagse chipproductietechnologie – iets dat een soepele overgang van de huidige computermachinerie naar het volgende niveau mogelijk zou kunnen maken.
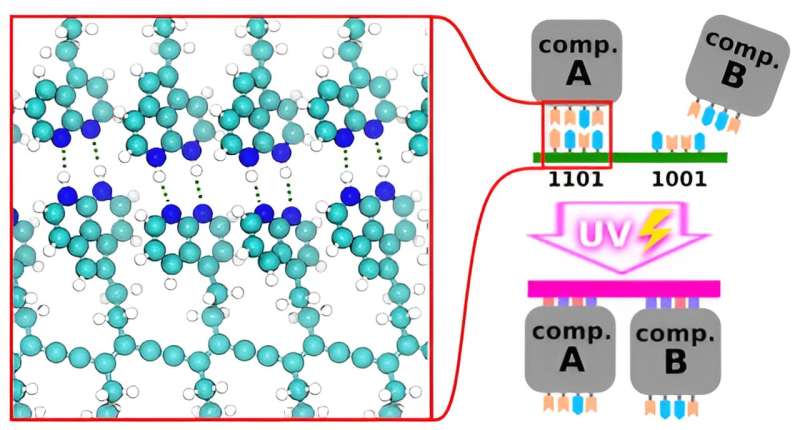
Om dit mogelijk te maken, stelden de onderzoekers voor om de suiker-fosfaten-skelet te vervangen door lichtgevoelige diacetyleen. Ze gebruikten gedetailleerde simulaties om te screenen op complementaire waterstofgebonden eindgroepen die de zelfassemblage op een rooster zouden aandrijven onder de omstandigheden die worden gebruikt bij de productie van chips.
Diacetyleenderivaten werden als ruggengraat gebruikt omdat ze onder deze omstandigheden efficiënt kunnen polymeriseren wanneer ze worden geprikkeld door UV-licht of elektroneninjectie, en eenheden analoog aan DNA / RNA-basen (de “letters” van de genetische code) werden in silico onderzocht als de eindgroepen. het assembleren van componenten in de beoogde vormen brengen.
Het doel was om complementaire paren te vinden, waarbij twee eenheden op betrouwbare wijze aan elkaar binden en niet aan andere eenheden. Deze eigenschap, opnieuw analoog aan hoe het DNA werkt, zou het creëren van deterministische complexe schakelpatronen mogelijk maken. De onderzoekers ontdekten dat eenheden met pure waterstofdonor-eindgroepen bijzonder geschikt waren. Er werden zestien veelbelovende kandidaat-eenheden gevonden, die de weg vrijmaakten voor experimenteel onderzoek en uiteindelijke industriële toepassingen.
De resultaten hebben interessante implicaties voor DNA-computergebruik en kunstmatige DNA-analogen. De meest haalbare vierletterige alfabetten die bij de screening werden gevonden, kwamen voor in een zeer smal gebied met bindingsenergieën van 15-25 kcal / mol, en waren allemaal afhankelijk van een kleine subset van de geteste eindgroepen.
Hoewel slechts een kleine subset van de mogelijke letterruimte met hoge nauwkeurigheid kon worden getest, suggereert dit dat het DNA-alfabet mogelijk niet alleen het resultaat is van een ‘ongeluk dat bevroren is in de tijd’, maar een stabiele en energetisch gunstige optie zou kunnen zijn geweest. In de geteste ruimte werden geen alfabetten van zes letters gevonden, maar nieuwe selectiviteitsmechanismen en niet-covalente bindingen anders dan waterstofbruggen (zoals halogeenbindingen) zouden dit mogelijk mogelijk kunnen maken. Op een vergelijkbare manier zouden de mogelijkheden van therapeutische en farmaceutische DNA-analogen kunnen worden getest.
Dit werk zal de synthetische beschikbaarheid van de moleculen verder verbeteren en experimentele beperkingen overwinnen. Hoewel de meesten van ons dit waarschijnlijk lezen over machines die afhankelijk zijn van op silicium gebaseerde transistors, zouden we binnenkort soepel kunnen overstappen op machines die gedeeltelijk gebruik maken van moleculaire nano-elektronica. Dit werk vertegenwoordigt een nieuwe stap in de richting van een dergelijke toekomst.
Meer informatie:
Mithun Manikandan et al., Computationeel ontwerp van lichtgevoelige polymeersjablonen om moleculaire nanofabricage aan te sturen, ACS Nano (2024). DOI: 10.1021/acsnano.3c10575
Tijdschriftinformatie:
ACS Nano
Geleverd door de Tsjechische Academie van Wetenschappen