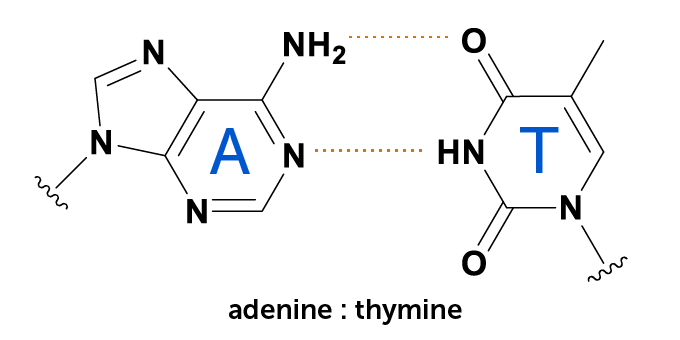
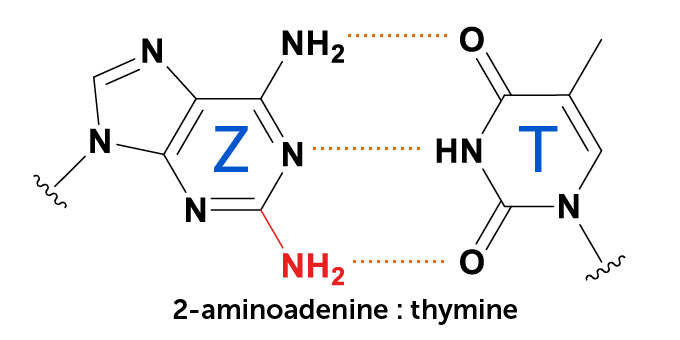
DNA heeft vier bouwstenen: A, C, T en G. Maar sommige bacteriofagen ruilen A voor Z
Sommige bacteriën infecteren virussen van de Siphoviridae (links) en Podoviridae (rechts) families die hier worden geïllustreerd, gebruiken een andere DNA-letter, Z, in plaats van de standaard A.
Sommige bacteriedodende virussen spellen hun genetische instructies in een ander DNA-alfabet.
Meer dan 40 jaar geleden meldden wetenschappers in Rusland dat een type bacteriofaag genaamd cyanofaag S-2L de DNA-bouwsteen adenine, algemeen bekend als A, vervangt door 2-aminoadenine, genaamd Z. Maar niemand wist hoe de faag van A ging. tot Z, of waarom.
Na decennia van afvragen hebben twee onafhankelijke groepen wetenschappers ontdekt hoe de virussen maken en bouwen Z in hun genetische instructies, en een reden waarom ze het doen, rapporteren de teams in drie onderzoeken op 30 april Wetenschap.
De bevindingen hebben implicaties voor het ontstaan van leven op aarde, de zoektocht naar leven op andere planeten en meerdere potentiële toepassingen in de biomedische geneeskunde, synthetische biologie, materiaalwetenschappen en informatica, zegt Farren Isaacs, een moleculaire en synthetische bioloog aan de Yale University die co-auteur was van een commentaar in hetzelfde nummer van Wetenschap. “Het is echt een fundamentele ontdekking.”
In de jaren negentig was Philippe Marlière, destijds xenobioloog aan het Pasteur Instituut in Parijs, “op zoek naar voorbeelden die afwijken van het leven zoals wij dat kennen”, toen hij de Russische studie uit 1977 tegenkwam waarin de cyanofaag met het ongebruikelijke DNA werd beschreven. Nadat ze een monster van het virus hadden gekregen, ontcijferden Marlière en collega’s de volledige set genetische instructies of het genoom van de faag.
In het genoom van het virus vonden de onderzoekers instructies voor het bouwen van een enzym, PurZ genaamd, dat de eerste stap zou kunnen uitvoeren om Z te maken – ook wel bekend als diaminopurine. Het Pasteur Instituut heeft in 2003 op naam van Marlière octrooi aangevraagd op het enzym.
Met het enzym in de hand “werd het glashelder hoe Z werd gemaakt, maar dat deden we niet [do] alle experimenten om te bewijzen dat we gelijk hadden ”, zegt Marlière, nu voorzitter van het Europese syndicaat van synthetische wetenschappers en industriëlen in Berlijn. Het project werd om verschillende redenen stopgezet.
De onderzoekers publiceerden hun bevindingen tot nu toe niet, deels omdat PurZ niet het enzym was waar Marlière naar op zoek was. In plaats daarvan zegt hij dat hij had gehoopt een ander enzym te vinden, een polymerase dat adenine zou afstoten en in plaats daarvan DNA zou bouwen met Z in plaats daarvan. “Ik was heel erg teleurgesteld”, zegt hij, “omdat het polymerase waar ik naar verlangde niet in die faag kon worden gedetecteerd.”
Inderdaad, het polymerase van deze faag is niet wat hij zocht. Marlière’s medewerker Pierre Alexandre Kaminski en collega’s ontdekten dat de polymerase van cyanofaag S-2L niet kieskeurig is over het gebruik van A of Z. In plaats daarvan, een andere virale Het enzym DatZ breekt de bouwstenen van adenine af, waardoor de polymerase geen andere keus heeft dan Z te gebruiken, Kaminski, een biochemicus bij het Pasteur Institute, en collega’s rapporteren 23 april in Nature Communications.
Marlière zocht periodiek genetische databases naar andere fagen die PurZ hebben en mogelijk het ongrijpbare kieskeurige polymerase bevatten. Dan, ongeveer vier jaar geleden, zegt hij: “Ik heb resultaten behaald. Ding, ding, ding! En ik heb er niet één gekregen. Ik heb er 12. En bingo, pal naast dit PurZ-gen, was, raad eens, een polymerase-gen. Aha! “
De Siphoviridae bacteriofagen die een grote verscheidenheid aan bacteriën infecteren, hebben allemaal versies van de polymerase, DpoZ genaamd, die bij voorkeur Z in plaats van A in het DNA van de virussen invoegen, rapporteren de onderzoekers. Marlière heeft patent aangevraagd op het enzym.
Het alternatieve alfabet kan veel breder worden gebruikt dan eerder werd gedacht, zegt Huimin Zhao, een synthetisch bioloog aan de Universiteit van Illinois in Urbana-Champaign. Hij hoorde een paar jaar geleden voor het eerst over de bacteriofaag die Z-bevattend DNA gebruikt op een etentje, herinnert hij zich. Niet wetende dat de Franse wetenschappers nog aan de puzzel werkten, zocht hij ook databases en vond 60 bacteriofagen die PurZ bevatten, inclusief fagen van beide Siphoviridae en Podoviridae gezinnen. Zijn team werkte ook de biochemische route uit die de fagen gebruiken om Z te maken en op te nemen, en vond enzymen die A. afbreken.
Alleen omdat de fagen de enzymen hebben, gebruiken ze niet noodzakelijk Z in hun DNA. Dus kozen Zhao en collega’s in China een faag genaamd SH-Ab 15497 die infecteert Acinetobacter bacteriën, en bevestigde dat het DNA-alfabet ook Z heeft in plaats van A, meldt zijn team.
Vervangen zoals bij Zs
Waarom fagen zich zouden bezighouden met het onconventionele DNA was nog niet bekend. Een hypothese is dat het vervangen van A door Z een tegenmaatregel is tegen bacteriële afweerenzymen, ook wel bekend als restrictie-enzymen, die DNA van binnendringende fagen fijnhakken. Zulke enzymen hebben moeite met het herkennen en knippen van DNA dat Z-basen bevat, vonden Zhao en collega’s. “De faag probeert te voorkomen dat hij door de gastheer wordt vernietigd”, zegt hij. “Dit is echt een beschermingsmechanisme voor de faag.”
Het maakt ook deel uit van een nooit eindigende wapenwedloop tussen fagen en bacteriën, zegt Steven Benner, een chemicus en astrobioloog bij de Foundation for Applied Molecular Evolution in Alachua, Florida. daarbuiten zijn. “We hebben deze levensvorm op aarde over het hoofd gezien omdat onze moleculaire hulpmiddelen ons niet in staat stelden ernaar te zoeken”, zegt hij. “Wat deze jongens hebben gedaan, is een hele biosfeer ontdekken die ontbrak in onze inventaris.”
Het is de vraag of Z-bevattende fagen nieuwe vormen van leven zijn (om nog maar te zwijgen van de voortdurende discussie over de vraag of virussen leven), zegt Floyd Romesberg, een synthetisch bioloog bij het wereldwijde farmaceutische en biotechnologiebedrijf Sanofi in La Jolla, Californië. opent nieuwe mogelijkheden, zegt hij, voor wat het leven is, was en zou kunnen worden.
‘Het leven is niet precies wat we dachten dat het was. Het leven hoeft geen GTAC te zijn ”, zegt hij, verwijzend naar de vier letters van het standaard DNA-alfabet. “Wat er staat, is dat het leven diverser kan zijn.”
Dat besef zou de zoektocht naar leven op andere planeten kunnen beïnvloeden (SN: 18-4-2016). Wetenschappers gaan er vaak van uit dat ze moeten zoeken naar guanine, thymine, adenine en cytosine, de basis van het DNA zoals we dat tot nu toe kenden. Maar misschien moeten onderzoekers in plaats daarvan zoeken naar 2-aminoadenine, de Z-base, zegt Benner.
Z vormt immers drie waterstofbruggen met thymine, in plaats van de twee waterstofbruggen die AT-basenparen bij elkaar houden. Dat maakt het Z-T-gepaarde DNA stabieler en mogelijk in staat om de warmere of zwaardere omstandigheden te weerstaan dan conventioneel DNA, zegt hij.

Met de extra stabiliteit zou je je kunnen afvragen waarom niet alle organismen op aarde Z gebruiken. Stabiliteit is niet alles, zegt Romesberg. DNA moet worden afgewikkeld en gesplitst om te worden gekopieerd. Dat is misschien moeilijker te doen met Z – T-basenparen. Z verandert ook de manier waarop DNA kromt en buigt, waardoor het misschien moeilijker wordt om in krappe ruimtes te stoppen zoals A-bevattend genetisch materiaal dat kan. Dat kan A aantrekkelijker maken voor andere organismen.
Of misschien was het gewoon een ongeluk dat A op de eerste plaats kwam. Zodra cellen die basis gingen gebruiken, zouden er te veel dingen moeten veranderen om volledig over te schakelen naar een andere basis, zegt Romesberg, die al jaren werkt om bacteriën exotische DNA-basen te laten inbouwen (SN: 7/5/14).
Cellen kunnen moeilijk worden verwisseld omdat er zoveel verschillende onderdelen zijn die moeten worden vervangen om een nieuwe DNA-basis te huisvesten. De uitgeklede genomen van virussen zijn flexibeler, zegt Romesberg: ze dragen minder machines rond omdat ze de gastheer het meeste werk laten doen. Zelfs de Z-faag doet slechts de eerste stap bij het maken van Z en vertrouwt op verschillende gastheerenzymen om het recept af te maken. Het is nog steeds niet bekend of cellulaire organismen ook Z in hun DNA kunnen schrijven.